 |
|


Revista Multidisciplinar Epistemología de las Ciencias
Volumen 2, Número 1, 2025, enero-marzo
GUÍA RÁPIDA DE ANÁLISIS GENÉTICOS BAJO MODELOS MIXTOS
CON WOMBAT
QUICK GUIDE TO GENETIC ANALYSIS FOR MIXED MODELS WITH
WOMBAT
José Raúl Pérez González
Venezuela

19 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Guía rápida de análisis genéticos bajo modelos mixtos con WOMBAT
Quick guide to genetic analysis for mixed models with WOMBAT
José Raúl Pérez González
josejrpg1995@email.com
https://orcid.org/0009-0007-2442-486X
Universidad politécnica territorial de
Maracaibo (UPTMA)
Venezuela
RESUMEN
La identificación de los mejores individuos se basa en la obtención de los valores genéticos (VG),
o mejores predictores lineales insesgados (BLUP) como también se les conoce, los cuales,
permiten la identificación de animales genéticamente superiores o inferiores en el rebaño. Un
programa informático muy utilizado a nivel mundial para calcular los VG es el WOMBAT, el cual
fue escrito en FORTRAN95 por la doctora Karin Meyer y lanzado al mercado en el año 2005. El
WOMBAT utiliza la metodología de modelos lineales mixtos bajo el método REML. Se utilizó una
base de datos de peso al destete en ganado vacuno, para mostrar el uso del WOMBAT en el
cálculo de los VG utilizando 3 modelos diferentes. Para los 3 modelos se encontraron resultados
diferentes para los VG. Se puede concluir que, el WOMBAT-REML, puede predecir los VG de
los animales para las características usando algoritmos eficientes bajo modelos mixtos, se
recomienda su uso para la evaluación genética de características de interés zootécnico en
poblaciones de animales.
Palabras clave: WOMBAT, valor genético, modelo animal, modelo mixto, REML.
20 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
ABSTRACT
The identification of the best individuals is based on obtaining the genetic values (GV), or best
linear unbiased predictors (BLUP) as they are also known, which allow the identification of
genetically superior or inferior animals in the herd. A computer program widely used worldwide to
calculate the GV is WOMBAT, which was written in FORTRAN95 by Dr. Karin Meyer and
launched on the market in 2005. WOMBAT uses the methodology of mixed linear models under
the REML method. A database of weaning weight in cattle was used to show the use of WOMBAT
in calculating the GV using 3 different models. For the 3 models different results were found for
the GV. It can be concluded that WOMBAT-REML can predict the GV of animals for the
characteristics using efficient algorithms under mixed models, its use is recommended for the
genetic evaluation of characteristics of zootechnical interest in animal populations.
keywords: WOMBAT, genetic value, animal model, mixed model, REML.
Recibido: 28 de diciembre 2024 | Aceptado: 28 de enero 2025
INTRODUCCIÓN
En la producción animal, es de interés aplicar programas de mejoramiento genético, con
la finalidad, de poder mejorar las características de interés económico en las poblaciones de
animales (Aranguren y col, 2007), para este fin, se tienen que, identificar los individuos
genéticamente superiores, para que, al reproducirlos, se mejoren los caracteres de interés en la
siguiente generación (Vilela, 2014). La identificación de los mejores individuos se basa en la
obtención de los valores genéticos (VG), conocidos como los mejores predictores lineales
insesgados o BLUP (Henderson, 1973), los cuales, permiten la identificación de animales
genéticamente superiores o inferiores en el rebaño (Blasco, 2021).
21 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Para calcular los VG, generalmente se utiliza el modelo lineal mixto (Littell y col, 2006),
el cual asume que, en el análisis se tendrán en cuenta tanto factores fijos como aleatorios
(Blasco, 2017). Un programa informático muy utilizado a nivel mundial para calcular los VG es
el WOMBAT (Meyer, 2007), el cual fue escrito en FORTRAN95 por la doctora Karin Meyer y
lanzado al mercado en el año 2005. El WOMBAT utiliza la metodología de modelos lineales
mixtos bajo el método REML (Patterson y Thompson, 1971) para calcular los VG y los
componentes de varianza (Henderson, 1953), estos últimos necesarios, para obtener los
parámetros genéticos (Vega, 1998). El programa puede utilizarse tanto en Windows de 32 o 64
bits o en entornos Linux.
Cálculo del valor genético usando varios tipos de información
Los VG predichos son predicciones obtenidas usando los modelos lineales (Searle y
col, 1992), según Castejón (2008), el modelo lineal es una expresión algebraica que, presenta
todos los factores de estudio, y modelo permite hacer predicciones tanto de los datos como de
los parámetros involucrados en el mismo. En el caso más simple, que se utiliza la información
del propio individuo (su dato propio), el modelo toma la siguiente forma (Pérez y Morales,
2023):
=
+
+
(1)
Donde
es el dato del animal,
es la media del rebaño,
es el VG de cada individuo
y
es el error residual. Bajo este modelo, se asume que
es un factor fijo y
y
son
aleatorios.
La expresión matemática que permite estimar el VG bajo el modelo 1 viene dado por
(Pérez, 2024):
=
2
(
(2)

22 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Donde
2
es la heredabilidad del carácter que se está estudiando. La correlación entre
y
para el modelo (1) viene dado por (Legates y Warwick, 1992):
=
2
(3)
Donde
es la correlación entre el VG predicho y el real.
Si se introduce un efecto fijo diferente de
el modelo toma la siguiente forma (Román y
Aranguren, 2014):
=
+
+
+
(4)
Donde
es el efecto del factor fijo. Para este modelo, las soluciones de
son
(Gutiérrez, 2010):
=
2
(
)
(5)
Donde
es la media para el nivel del efecto fijo donde el dato del animal está presente.
Si se quiere utilizar toda le genealogía en la predicción de los VG, es necesario
encontrar las soluciones de las ecuaciones normales de Henderson para los parámetros del
modelo, incluyendo la matriz de parentesco. Para la ecuación 4 las ecuaciones son (Elzo,
2012):
+
1
=
(6)
Donde X es una matriz de incidencia que relaciona los datos con los factores fijos, Z es
una matriz de incidencia que relaciona los datos con
,
1
es la inversa de la matriz de
parentesco,
es un vector de datos y
es un escalar que introduce
2
en las ecuaciones, el
cual toma la siguiente fórmula (Mrode y Thompson, 2005):
=
1
2
2
(7)
23 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
El error estándar de
en el caso general viene dado por (Aranguren y Román, 2014):
(
)
=
2
(8)
Donde
es el inverso de la diagonal de la matriz de coeficientes para los bloques de
animales y
2
es la varianza residual.
La correlación
para el caso general viene dado por (Solarte y col, 2024):
=
1
(9)
Estimación de los parámetros genéticos
Los parámetros genéticos se estiman usando los componentes de varianza, para el
caso del modelo reproductor (sire model), la fórmula es (Becker, 1986):
2
=
4
2
2
=
2
2
+
2
(10)
Donde
2
es la varianza aditiva,
2
es la varianza residual,
2
es la varianza fenotípica y
2
es la varianza entre reproductores.
Con el modelo animal, la heredabilidad puede estimarse directamente usando la
varianza aditiva y la residual (Fernández y col, 2021):
2
=
2
2
+
2
(11)
La repetibilidad viene dado por (Falconer, 2001):
=
2
+
2
2
(12)
Donde
2
es la varianza de ambiente permanente.

24 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Existen varios métodos para estimar los componentes de varianza (Sorensen y Gianola,
2002), en WOMBAT se utiliza el método de REML, el cual consiste en encontrar los parámetros
estimados a través de la maximización del logaritmo de la función de verosimilitud restringida,
la cual, es una función de los parámetros dados los datos. El logaritmo de la función de
verosimilitud en algebra de matrices viene dado por (Pérez y col, 2024):
(
)
=
|
|
|
1
|
0.5
2
ln
(13)
Donde V es la matriz de varianzas y covarianzas fenotípicas y
es la suma de
cuadrados residual generalizada. Como
2
ln
es una constante y desaparecer al derivar,
la expresión puede acomodarse como (Meyer, 1997):
(
)
=
0.5
+
|
|
+
|
1
|
+
(14)
Maximizar
(
)
es equivalente a minimizar -2Ln(L), y en algebra de matrices esta
expresión es (Meyer, 1989)
(
)
=
+
|
|
+
|
|
+
|
|
+
(15)
Donde C es la matriz de coeficientes de las ecuaciones normales de Henderson (Verde
y Yañez, 2014), R es una matriz de varianzas residual expresada como
=
2
y G es una matriz
de varianzas aditivas expresada como
=
2
.
En un modelo reproductor para alcanzar el máximo de
(
)
, si la data es balanceada,
puede derivarse
(
)
en función de los parámetros y resolver las ecuaciones lineales, pero si
la data es desbalanceada se tienen que aplicar métodos numéricos iterativos (Pérez, 2024).
Uno de estos métodos usados para maximizar
(
)
es el AI-REML que se traduce como
algoritmo de información promedio (Johnson y Thompson, 1995), otros métodos son los libre
de derivadas, como el POWEL y el SIMPLEX (Boldman y col, 1995), ambos incorporados en el
25 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
WOMBAT. En el caso del modelo animal, en todos los casos tienen que aplicarse métodos
numéricos iterativos, para encontrar los componentes de varianza.
Introducciones en WOMBAT
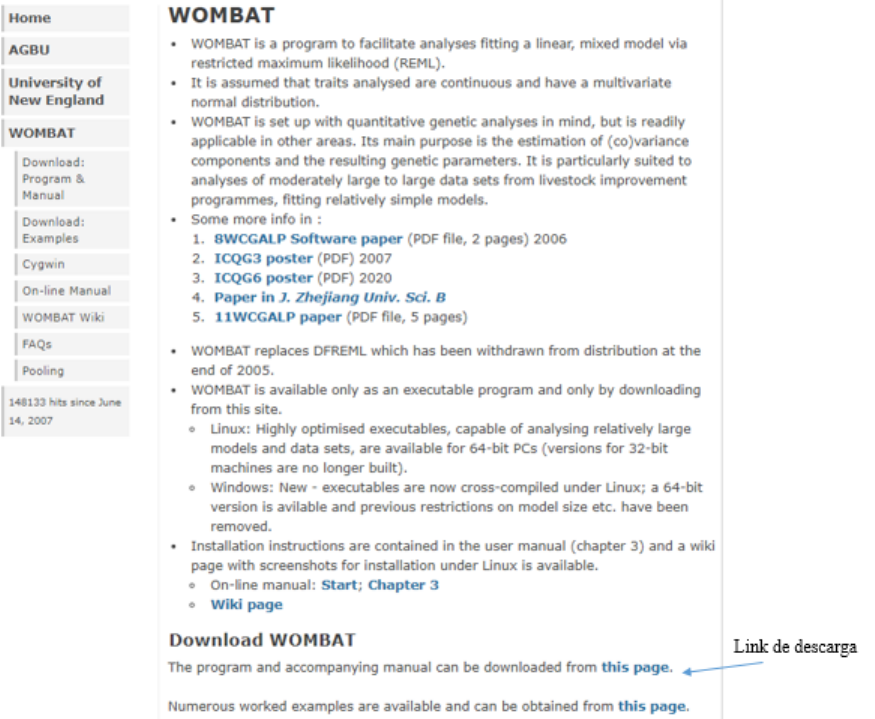
El ejecutable del programa WOMBAT puede descargarse gratuitamente para Linux y
Windows desde la página http://didgeridoo.une.edu.au/km/wombat.php. En la figura 1 se
muestra una imagen de la página de WOMBAT indicando donde descargar el programa:
Figura 1
Página para descargar WOMBAT.
26 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Una vez ingresado al link, existen varias opciones de ejecutables WOMBAT para Linux
y Windows para 32 y 64 bit.
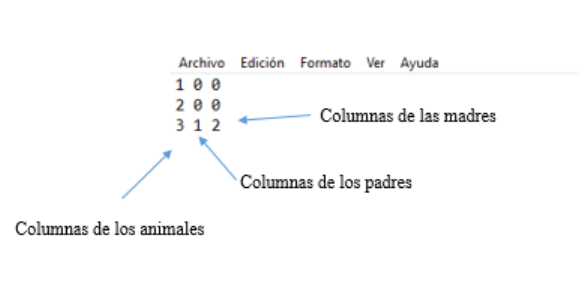
Para ejecutar WOMBAT es necesario crear tres archivos, el primero es el archivo
genealógico, en el cual se escribe la identificación numérica de los animales y su genealogía
(identificación de padre y madre). Este archivo debe tener una extensión .PED, y requiere crear
3 columnas, la primera para la identificación de los animales, la segunda para la identificación
de los padres y la tercera para las madres; estas columnas para los animales, padres y madres
se escriben de izquierda a derecha respectivamente. En el caso de que un animal no se
conozca algún padre se coloca cero (0), por último, el número de los padres siempre tiene que
ser menor que el de sus hijos. En la figura 2 se muestra un ejemplo de un archivo genealógico.
Figura 2
Ejemplo archivo genealógico.
En este ejemplo, podemos ver que tenemos tres animales (1,2 y 3), los animales 1 y 2
no tienen padres conocidos y el animal 3 tiene como padres a los animales 1 y 2.
El segundo archivo es el archivo de datos, el cual incluye la identificación de los
animales y todos los factores involucrados en el modelo estadístico. El archivo de datos tiene
una extensión .DAT y requiere estrictamente que todos los factores involucrados en el análisis
sean colocados en el archivo de forma numérica, por ejemplo, si deseamos ajustar un efecto
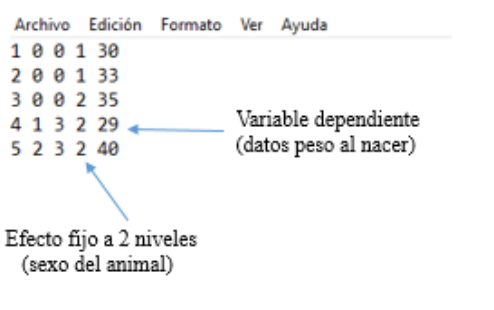
27 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
fijo con 2 niveles, al primer nivel del efecto fijo se le puede colocar el valor 1, y al segundo nivel
un valor 2. En la figura 3 se muestra un ejemplo de un archivo de datos:
Figura 3
Ejemplo archivo de datos
En la figura 3, las tres primeras columnas son de la identificación de los animales, los
padres y las madres, la cuarta columna es para el efecto fijo sexo del animal, el cual, está a dos
niveles; el valor es 1 para los machos y el 2 para las hembras, por último, la quinta columna
son los datos de la variable dependiente el cual es el peso al nacer de cada animal.
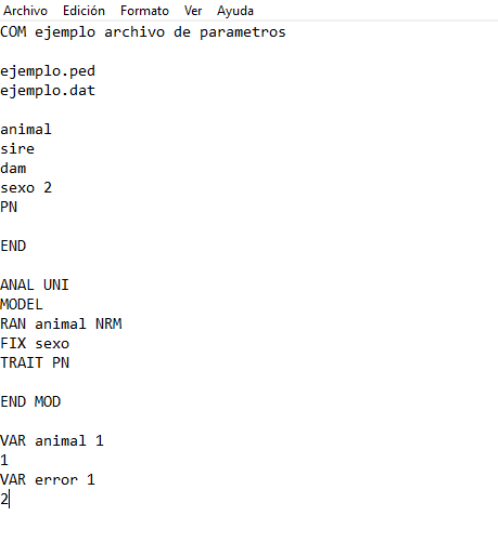
El tercer archivo es el archivo de parámetros, el cual proporciona toda la información
sobre los archivos de datos, genealógico, y el modelo de análisis que se va a ajustar. Este
archivo tiene una extensión .PAR y tiene sentencias específicas que se pueden consultar en el
manual. En este texto se explican las instrucciones principales del archivo de parámetros,
tomando como ejemplo el archivo de datos de la figura 3. En la figura 4 se muestra el archivo
de parámetros para la figura 3:
Figura 4
Ejemplo archivo de parámetros
28 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
La primera fila con el carácter COM indica un comentario, luego las siguientes dos filas
son los nombres del archivo genealógico y de datos con su extensión, luego las variables que
están en el archivo de datos con el número de niveles para el factor fijo, END indica la
finalización de la entrada de variables, ANAL UNI indica que se quiere usar un modelo
univariado, RAN indica que la variable animal es aleatoria y FIX indica que el factor sexo es fijo,
TRAIT indica que el peso al nacer (PN) es la variable respuesta, END MOD indica que el
modelo finalizo y por último se dan los valores iniciales para los componentes de varianza.
Para utilizar el WOMBAT es requerido guardar en una carpeta los archivos de
parámetro, genealógico, de datos y el ejecutable de WOMBAT, luego desde el símbolo del
sistema se ubica la carpeta y se coloca la palabra wombat y luego el nombre del archivo de
parámetros con su extensión. Entre la palabra wombat y el nombre del archivo de parámetros,
puede colocarse algún código que indique al programa un paso a tomar, por ejemplo, la opción
–blup indica al wombat que calcule directamente los VG usando los valores iniciales de los
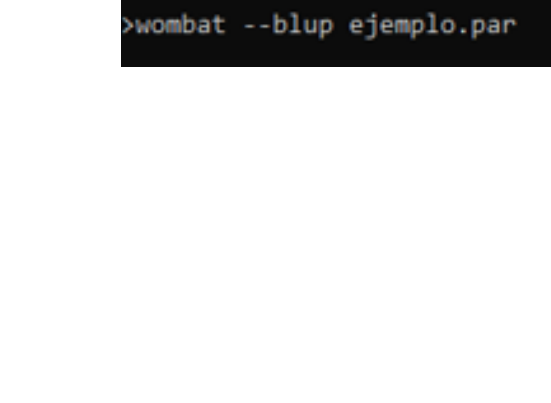
29 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
componentes de varianza. En la figura 5 se muestra como colocar los caracteres para usar
WOMBAT:
Figura 5
Caracteres para el uso del WOMBAT
En la figura 5 se puede ver la secuencia para utilizar WOMBAT, primeramente, se
coloca wombat, luego la opción a utilizar y por último el nombre del archivo de parámetros con
su extensión.
Otros programas para calcular los VG bajo modelos mixtos se pueden encontrar en la
red, algunos de ellos son el MTFREML (Boldman y col, 1995), Echidna MMS (Gilmour, 2021) y
el BLUPF90 (Misztal y col, 2014; Lourenco y col, 2022). Aunque no es el objetivo de este trabajo
hacer investigaciones entre programas, los citados anteriormente al igual que el wombat, son
programas muy utilizados a nivel internacional para realizar las evaluaciones genéticas en
animales de interés zootécnico.
METODOLOGIA
Desarrollado con una metodología cuantitativa basada en el análisis de datos utilizando
modelos estadísticos, se utilizó una base de datos de peso al destete (PD) en ganado vacuno,
para mostrar el uso del WOMBAT en el cálculo de los VG, los datos del ejemplo se pueden
apreciar en la tabla 1:

30 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Tabla 1
Datos del ejemplo de PD
Animal
Padre
Madre
Sexo
y (PD)
1
0
0
M
205
2
0
0
M
200
3
0
0
H
195
4
1
0
H
188
5
2
0
H
170
Se puede aprecia en la tabla 1 que se evaluarán 5 animales, donde el 1 y 2 son padres
de los animales 4 y 5, no existen madres conocidas, se tienen 2 animales machos (1 y 2), 3
hembras (3, 4 y 5) y los datos del PD. En el ejemplo se asumió que
2
=
0.50
y se usaron
varios modelos estadísticos lineales. EN la tabla 2 se muestran los modelos ajustados:
Tabla 2
Modelos ajustados para ejemplo
Numero
Modelo
Genealogía
Archivo de
parámetros
1
=
+
+
+
RAN animal NRM
TRAIT PD
2
=
+
+
+
+
RAN animal NRM
FIX sexo 2
TRAIT PD
3
=
+
+
+
+
1
RAN animal NRM
FIX sexo 2

31 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
TRAIT PD
Para los 3 modelos se utilizó la opción –blup del WOMBAT para predecir los VG usando
2
=
1
y
2
=
1.
RESULTADOS
La predicción del VG con sus EE y correlaciones para los 5 animales usando los
modelos 1 y 2 se presentan en la tabla 3:
Tabla 3
Predicciones del VG, EE y correlaciones para los modelos 1 y 2.
Modelo 1
Animal
VG
EE
Correlación
1
1
=
0.5
(
205
191.6
)
=
6.7
0.5
=
0.707106
0.5
=
0.707106
2
2
=
0.5
(
200
191.6
)
=
4.2
0.5
=
0.707106
0.5
=
0.707106
3
3
=
0.5
(
195
191.6
)
=
1.7
0.5
=
0.707106
0.5
=
0.707106
4
4
=
0.5
(
188
191.6
)
=
1.8
0.5
=
0.707106
0.5
=
0.707106
5
5
=
0.5
(
170
191.6
)
=
10.8
0.5
=
0.707106
0.5
=
0.707106
Modelo 2
1
1
=
0.5
(
205
202.5
)
=
1.25
0.75
=
0.866025
1
0.75
=
0.5
2
2
=
0.5
(
200
202.5
)
=
1.25
0.75
=
0.866025
1
0.75
=
0.5
3
3
=
0.5
(
195
184.333
)
=
5.3335
0.66666
=
0.81649
1
0.666
=
0.577
4
4
=
0.5
(
188
184.333
)
=
1.8335
0.66666
=
0.81649
1
0.666
=
0.577

32 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
5
5
=
0.5
(
170
184.333
)
=
7.1665
0.66666
=
0.81649
1
0.666
=
0.577
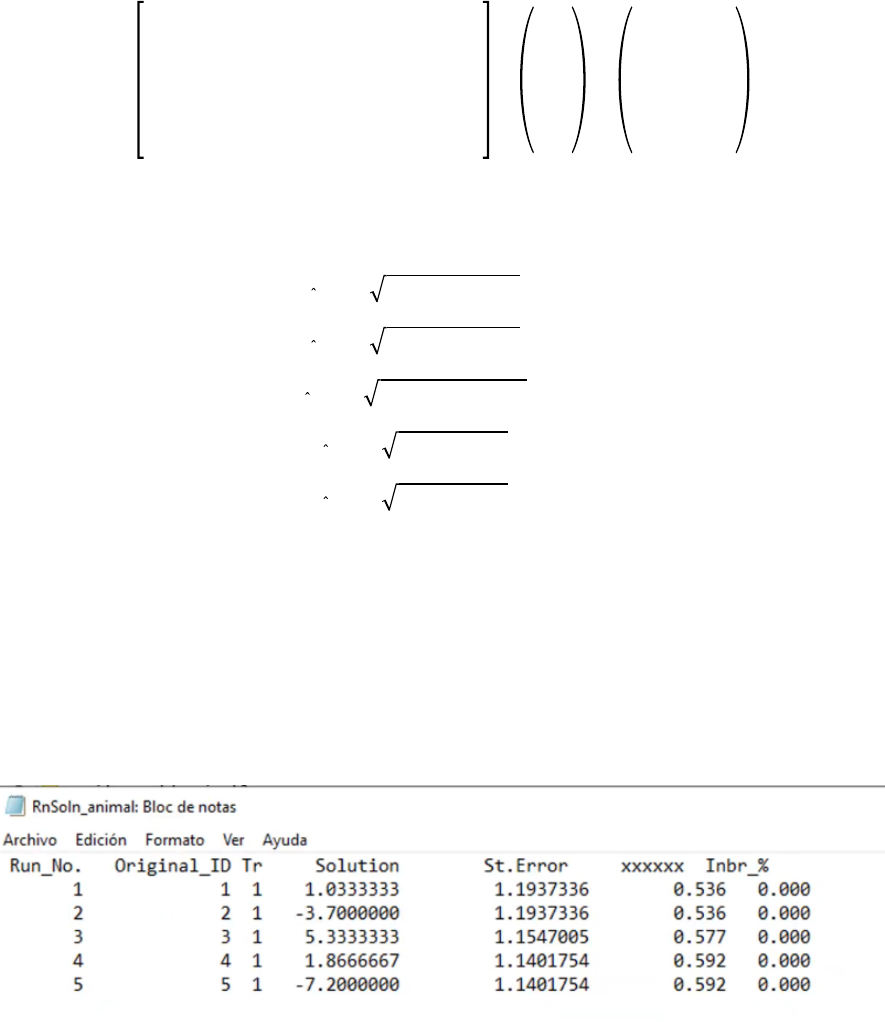
En la figura 6 se presenta la salida del WOMBAT para el modelo 1.
Figura 6
Salida WOMBAT modelo 1
Se puede apreciar en la figura 6 resultados idénticos a los calculados manualmente,
adicionalmente el WOMBAT calcula el porcentaje de consanguinidad de cada individuo, pero
en este caso, al no tener genealogías de los animales, el coeficiente es 0. Bajo este modelo el
mejor animal es el 1 (+6.7) y el peor es el 5 (-10.8). En la figura 7 se presenta la salida para el
modelo 2:
Figura 7
Salida WOMBAT modelo 2
33 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Tanto los VG, correlaciones y los errores estándares son idénticos a los obtenidos de
manera manual, bajo este enfoque el mejor animal es el 3 (+5.333) y el peor sigue siendo el 5
(-7.166) aunque su VG mejoró. Los cambios en los VG se deben a la introducción del factor
sexo en el modelo, por lo tanto, se elimina la variación ocasionada por el factor (sexo) en la
predicción de los VG.
Para el modelo 3 se necesita calcular la inversa de la matriz de parentesco, para el
pedigree de la tabla 1, la matriz
1
usando la regla de Henderson (Gutiérrez, 2010) viene
dada por:
1
=
1
+
1
/
3
0
0
2
/
3
0
0
1
+
1
/
3
0
0
2
/
3
0
2
/
3
0
0
0
2
/
3
1
0
0
0
4
/
3
0
0
0
4
/
3
4
/
3
0
0
2
/
3
0
0
4
/
3
0
0
2
/
3
0
2
/
3
0
0
0
2
/
3
1
0
0
0
4
/
3
0
0
0
4
/
3
El escalar
para
2
=
0.50
es:
=
1
0.5
0.5
=
1
Las ecuaciones normales de Henderson quedan definidas como:
2
0
0
3
1
1
0
0
0
0
0
1
1
1
1 0
1 0
0 1
0 1
0 1
7
/
3
0
0
2
/
3
0
0
7
/
3
0
0
2
/
3
0
2
/
3
0
0
0
2
/
3
2
0
0
0
7
/
3
0
0
0
7
/
3
1
2
1
2
3
4
5
=
405
553
205
200
195
188
170
Y la solución de este sistema de ecuaciones viene dada por:
34 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
2
0
0
3
1
1
0
0
0
0
0
1
1
1
1 0
1 0
0 1
0 1
0 1
7
/
3
0
0
2
/
3
0
0
7
/
3
0
0
2
/
3
0
2
/
3
0
0
0
2
/
3
2
0
0
0
7
/
3
0
0
0
7
/
3
1
405
553
205
200
195
188
170
=
203.83333
184.33333
1.03333
3.7
5.33333
1.86666
7.2
Y las correlaciones son:
1
1
=
1
0.7125(1)
=
0.53
2
2
=
1
0.7125(1)
=
0.53
3
3
=
1
0.66666
(
1
)
=
0.57
4
4
=
1
0.65(1)
=
0.59
5
5
=
1
0.65(1)
=
0.59
En la figura 8 se presentan la salida del WOMBAT para el modelo 3:
Figura 8
Salida del Wombat modelo 3
Se pueden apreciar resultados idénticos a los mostrados de manera manual, en este
caso, el peor animal posee un VG de -7.2 kg y el mejor un VG de 5.33 kg.

35 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
DISCUSIÓN
En varios trabajos puede encontrarse como construir las ecuaciones normales de
Henderson del modelo mixto, según Aranguren y Román (2014), las ecuaciones se construyen
de igual forma que, las ecuaciones normales de mínimos cuadrados, con la diferencia de que,
se añade
1
en las ecuaciones y para el modelo 1 las incógnitas pueden despejarse en las
ecuaciones, obteniendo para cada animal la siguiente expresión:
=
1
+
(16)
Tomando la expresión (16) y sustituyendo los valores para el animal 1 se obtiene
1
=
205
191.6
1
1
=
6.7
, un valor que, es idéntico al encontrado en este trabajo usando la fórmula
(2)
.
De igual forma, para el modelo 2, se puede despejar
encontrando la siguiente
solución:
=
1
+
(17)
Tomando los valores del animal 1 usando el modelo 2, se encuentra que, la solución del
VG es
1
=
205
202.5
1
1
=
1.25
un valor idéntico al encontrado en este trabajo usando la fórmula
(
5
)
.
Se puede encontrar otra fórmula para
despejando cualquier VG de la fila de
:
=
(18)
Donde
es la suma de los VG diferentes del animal que, se quiere evaluar. Para
el animal 1 la solución es
1
=
958
(
4..2
+
1.7
1.8
10.8
)
5(191.6)
=
6.7
el cual es un
resultado idéntico al encontrado en este trabajo. Las soluciones usando la ecuación (18)
36 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
aunque matemáticamente son correctas, en la práctica son muy poco usadas, ya que requieren
el VG de todos los animales evaluados previamente, cosa que, por lo general no se conoce.
Para el modelo animal con relaciones de parentesco, como se trabaja en el modelo 3,
se pueden encontrar fórmulas con resultados equivalentes a los obtenidos en este trabajo.
Mrode y Thompson (2005) explican que, en un modelo animal, las soluciones de las
ecuaciones normales de Henderson son equivalentes a la siguiente expresión matricial:
+
1
=
(
)
(19)
Por lo tanto, para los datos del modelo 3, las soluciones son:
=
7
/
3
0
0
2
/
3
0
0
7
/
3
0
0
2
/
3
0
2
/
3
0
0
0
2
/
3
2
0
0
0
7
/
3
0
0
0
7
/
3
1
1
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
1
0
0
0
1
0
0
0
1
205
200
195
188
170
1
0
1
0
0
0
0
1
1
1
203.83333
184.33333
=
1.03333
3.7
5.33333
1.86666
7.2
Estos resultados son idénticos a los obtenidos usando la solución directa de las
ecuaciones normales de Henderson, sin embargo, para encontrar estas soluciones se necesita
el valor de las estimaciones de los efectos fijos, por lo tanto, en la práctica no suele ser muy
usada.
CONCLUSIONES
Se puede concluir que, el WOMBAT-REML, puede predecir los VG de los animales para
las características usando algoritmos eficientes bajo modelos mixtos, se recomienda su uso
para la evaluación genética de características de interés zootécnico en poblaciones de
animales.
REFERENCIAS
Aranguren, A., & Román, R. (2014). El modelo animal simple: una metodología para los
genetistas. Logros & Desafíos de la Ganadería Doble Propósito, GIRAZ, 120-136.

37 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Aranguren, A., Román, R., Villasmil, Y., & Yañez, F. (2007). Evaluación genética de la
ganadería mestiza doble propósito en Venezuela. Archivos Latinoamericanos de
Producción Animal, 15(1), 241-250.
Becker, W. (1986). Manual de genética cuantitativa. Academic Enterprises.
Blasco, A. (2017). Bayesian data analysis for animal scientists: The basics. Springer.
Blasco, A. (2021). Mejora genética animal. Editorial Síntesis.
Boldman, K. G., Kriese, L. A., Van Vleck, L. D., Van Tassell, C. P., & Kachman, S. D. (1995). A
manual for use of MTDFREML: A set of programs to obtain estimates of variances and
covariances [Draft]. U.S. Department of Agriculture, Agricultural Research Service.
Castejón, O. (2008). Diseño y análisis de experimentos con Statistix. Colección de Textos
Universitarios. Ediciones del Vicerrectorado Académico.
Elzo, M., & Garay, O. (2012). Modelación aplicada a las ciencias animales: II. Evaluaciones
genéticas. Editorial Biogénesis.
Falconer, D. (2001). Introducción a la genética cuantitativa. Longman.
Fernández, N., Herrera, J. C., Pérez, N. G., Doria, M. R., Mestra, L. V., & Lucero, C. (2021).
Heredabilidades para características de crecimiento a través de los años en la raza
Blanco Orejinegro. Revista de Investigaciones Veterinarias del Perú, 32(5).
Gilmour, A. R. (2021). Echidna Mixed Model Software. Recuperado de www.EchidnaMMS.org.
Gutiérrez, P. (2010). Iniciación a la valoración genética animal: Metodología adaptada al EEES.
Editorial Complutense.
Henderson, C. (1953). Estimation of variance and covariance components. Biometrics, 9(2),
226-252.
Henderson, C. (1973). Sire evaluation and genetic trends. En Proceedings of the Animal
Breeding Genetics Symposium in Honor of J.L. Lush (pp. 10-41). American Society of
Animal Science. https://doi.org/10.1093/ansci/1973.Symposium.10
38 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Johnson, D., & Thompson, R. (1995). Restricted maximum likelihood estimation of variance
components for univariate animal models using sparse matrix techniques and average
information. Journal of Dairy Science, 78, 449-456.
Legates, E., & Warwick, J. (1992). Cría y mejora del ganado. Interamericana McGraw-Hill.
Littell, R., Milliken, G., Stroup, W., Wolfinger, R., & Schabenberger, O. (2006). SAS for mixed
models. SAS Press.
Lourenco, D., Tsuruta, S., Masuda, Y., Bermann, M., Legarra, A., & Misztal, I. (2022).
Actualizaciones recientes en el paquete de software BLUPF90. Congreso Mundial de
Genética Aplicada a la Producción Ganadera.
Meyer, K. (1989). Restricted maximum likelihood to estimate variance components for animal
models with several random effects using a derivative-free algorithm. Genetics Selection
Evolution, 21, 317-340.
Meyer, K. (1997). An ‘average information’ restricted maximum likelihood algorithm for
estimating reduced rank genetic matrices or covariance functions for animal models with
equal design matrices. Genetics Selection Evolution, 29, 97-116.
Meyer, K. (2007). WOMBAT: A tool for mixed model analyses in quantitative genetics by
restricted maximum likelihood (REML). Journal of Zhejiang University Science B, 8(11),
815-821.
Misztal, I., Tsuruta, S., Lourenco, D. A. L., Aguilar, I., Legarra, A., & Vitezica, Z. (2014). Manual
para la familia de programas BLUPF90.
Mrode, R., & Thompson, P. (2005). Linear models for the prediction of animal breeding values
(2ª ed.). CABI Publishing.
Patterson, H., & Thompson, R. (1971). Recovery of inter-block information when block sizes are
unequal. Biometrika, 58, 545-554.
Pérez, J. (2024). Estadística aplicada al mejoramiento genético animal. Fondo Editorial
Universidad Rafael Urdaneta.
39 Revista Multidisciplinar Epistemología de las Ciencias | vol. 2, núm. 1, 2025 | enero-marzo
Pérez, J., Jiménez, E., & Morales, D. (2024). Repetibilidad del intervalo entre parto en ganado
Carora en Venezuela. RECITIUTM, 10(2).
Pérez, J., & Morales, D. (2023). Theory of estimation of parameters and genetic values under
mixed models. International Journal of Avian & Wildlife Biology, 8(1), 27-33.
https://doi.org/10.15406/ijawb.2024.08.00210
Román, R., & Aranguren, A. (2014). Evaluación genética de reproductores: Logros y desafíos.
GIRAZ.
Searle, S. R., Casella, G., & McCulloch, C. E. (1992). Variance components. Wiley.
Solarte, C., Martínez, C., & Cerón, M. (2024). Modelos lineales para evaluación genética en
animales. Editorial UTP.
Sorensen, D., & Gianola, D. (2002). Likelihood, Bayesian, and MCMC methods in quantitative
genetics. Springer.
Vega, P. (1998). Introducción a la teoría de genética cuantitativa con especial referencia al
mejoramiento de plantas. UCV-Ediciones de la Biblioteca.
Verde, O., & Yañez, F. (2014). Modelos estadísticos de evaluación genética. Logros & Desafíos
de la Ganadería Doble Propósito, GIRAZ, 107-119.
Vilela, J. (2014). Mejoramiento genético animal en animales domésticos. Editorial Macro. Lima,
Perú.